Международный неврологический журнал Том 20, №8 2024
Вернуться к номеру
Порівняльна ефективність методів генетичної діагностики в дітей з розладами нервової системи в Україні
Авторы: Кирилова Л.Г. (1), Мірошников О.О. (1), Харитонов В.І. (2), Доленко О.О. (3), Бадюк В.М. (3), Дюкарева-Бездєнєжних С.В. (1)
(1) - ДУ «Всеукраїнський центр материнства та дитинства НАМН України», м. Київ, Україна
(2) - КНП «Клінічна лікарня «Психіатрія», м. Київ, Україна
(3) - ТОВ «Ультрагеном», м. Київ, Україна
Рубрики: Неврология
Разделы: Клинические исследования
Версия для печати
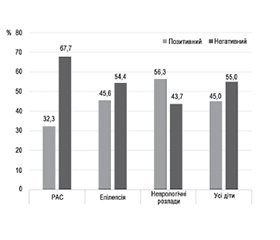
Актуальність. У сучасному суспільстві спостерігається значне зростання кількості нейропсихічних розладів у дітей. Важливу роль у діагностуванні захворювань нервової системи та порушень нейророзвитку відіграють сучасні методи генетичного тестування. Мета: проаналізувати результати генетичних досліджень у дітей з різними видами неврологічних розладів і оцінити ефективність цих методів для поліпшення підходів до діагностики. Матеріали та методи. У дослідження було включено 160 дітей віком 0–18 років (середній вік 6,7 року). Серед них 131 дитині було проведено повноекзомне секвенування (ПЕС), у тому числі 68 (51,9 %) — з епілепсією, 31 (23,7 %) — з розладами аутистичного спектра (РАС) і 32 (24,4 %) — з неврологічними розладами. Хромосомний мікроматричний аналіз (ХМА) проведено 56 дітям. Серед них 27 дітям проведено як ПЕС, так і ХМА. Результати. Серед обстеженої методом ПЕС 131 дитини позитивний результат було отримано в 59 пацієнтів (45,0 %), у тому числі серед дітей з епілептичними нападами — у 31 пацієнта (45,6 %), серед дітей з РАС — у 10 пацієнтів (32,3 %), серед дітей з неврологічними розладами — у 18 пацієнтів (56,3 %). Методом ХМА позитивний результат було отримано у 18 дітей (32,1 %), у тому числі серед дітей з епілепсією — у 5 (31,3 %), у дітей з РАС — в 11 (30,6 %), у дітей з неврологічними розладами — у 2 (50,0 %). Серед обстежених методом ХМА дітей 27 (48,2 %) попередньо були обстежені методом ПЕС і отримали негативний результат. У даній когорті у 8 дітей (29,6 %) було виявлено мутації методом ХМА, натомість серед дітей, у яких ПЕС не проводилося, методом ХМА було виявлено мутації в 10 дітей (34,5 %). Висновки. ПЕС є найбільш ефективним методом при неврологічних розладах (56,3 % дітей) та епілепсії (45,6 % дітей). У цілому метод ПЕС показав більшу ефективність у загальній вибірці дітей (45,0 %) порівняно з ХМА (32,1 %). Метод ХМА показав більшу ефективність при неврологічних розладах (50,0 %). Серед дітей з негативним результатом ПЕС у 29,6 % виявлено патогенні варіанти методом ХМА. Дослідження виконано відповідно до принципів Гельсінської декларації. На проведення досліджень отримано інформовану згоду пацієнтів і їхніх батьків. Автори заявляють про відсутність конфлікту інтересів.
Background. In modern society, there is a significant increase in the number of neuropsychiatric disorders among children. Modern methods of genetic testing play an important role in diagnosing diseases of the nervous system and neurodevelopmental disorders. The purpose was to analyze the results of genetic studies in children with various neurological disorders and to evaluate their effectiveness in improving diagnostic approaches. Materials and methods. One hundred and sixty children aged 0–18 years (average age 6.7 years) were included in the study. Among them, 131 underwent whole-exome sequencing (WES): 68 (51.9 %) with epilepsy, 31 (23.7 %) with autism spectrum disorders (ASD), and 32 (24.4 %) with neurological disorders. Chromosomal microarray (CMA) was performed in 56 children, including 27 children who underwent both WES and CMA. Results. Among 131 children examined by WES, a positive result was obtained in 59 (45.0 %) cases: 31 patients (45.6 %) with epileptic seizures, 10 children (32.3 %) with ASD, 18 (56.3 %) with neurological disorders. A positive result was obtained by the CMA method in 18 children (32.1 %): 5 (31.3 %) with epilepsy, 11 (30.6 %) with ASD, 2 (50.0 %) with neurological disorders. Among the children examined by CMA, 27 (48.2 %) were previously examined using WES and received a negative result. In this cohort, 8 children (29.6 %) had mutations detected by the CMA method, whereas among children who did not undergo WES, mutations were detected by CMA in 10 cases (34.5 %). Conclusions. WES is the most effective method in neurological disorders (56.3 %) and epilepsy (45.6 % of children). In general, WES showed greater effectiveness in the total sample of children (45.0 %) compared to CMA (32.1 %). CMA showed higher effectiveness in neurological disorders (50.0 %). Among children with a negative WES result, 29.6 % had pathogenic variants detected by CMA. The research was carried out in accordance with the principles of the Declaration of Helsinki. Informed consent for the research was obtained from the patients and their parents. The authors declare no conflict of interest.
розлади нейророзвитку; епілепсія; генетичне тестування; розлади аутистичного спектра; повноекзомне секвенування; хромосомний мікроматричний аналіз
neurodevelopmental disorders; epilepsy; genetic testing; autism spectrum disorders; whole-exome sequencing; chromosomal microarray