Журнал «Здоровье ребенка» Том 16, №1, 2021
Вернуться к номеру
Світ мікроРНК гепатобіліарної системи
Авторы: Абатуров О.Є., Бабич В.Л.
ДЗ «Дніпропетровська медична академія Міністерства охорони здоров’я України», м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді наведений світ мікроРНК гепатобіліарної системи. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Було підкреслено, що мікроРНК у клітинах відіграють важливу роль у регуляції активності експресії генів і контролюють численні фізіологічні процеси, такі як обмін речовин, проліферація, диференціювання, апоптоз клітин. Відображена асоціація деяких захворювань зі зміною вмісту мікроРНК у периферійному руслі крові. У статті наведена коротка характеристика групи некодуючих РНК. Надана характеристика основних баз даних мікроРНК із відображенням електронних адрес. Для визначення мікроРНК використовують як традиційні методи, засновані на технології ампліфікації, так і нові методи детекції (секвенування нового покоління, електрохімічне детектування на основі посилення ферментатного сигналу, ідентифікація за допомогою лігування і застосування золотих наночастинок). Автори проводять порівняння різних методів детекції мікроРНК. Зазначено, що надекспресія або інгібування генерації специфічних мікроРНК супроводжуються порушеннями найважливіших функцій печінки і розвитком захворювань гепатобіліарної системи. Продемонстровано, що зміни деяких мікроРНК у сироватці крові або тканині печінки є високодіагностичними маркерами деяких захворювань печінки. Таким чином, ідентифікація зміни рівня презентабельності певних мікроРНК може дати цінну діагностичну інформацію практикуючому лікарю, а вплив на процеси утворення та матурації мікроРНК за допомогою лікарських засобів становить собою новий напрямок терапії широкого спектра захворювань. Особливий інтерес викликає сучасне уявлення про діагностичне значення мікроРНК при захворюваннях біліарного тракту в дітей та можливості медикаментозного управління активністю процесу їх генерації.
В научном обзоре представлен мир микроРНК гепатобилиарной системы. Для написания статьи осуществлялся поиск информации с использованием баз данных Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Было подчеркнуто, что микроРНК в клетках играют важную роль в регуляции активности экспрессии генов и контролируют многочисленные физиологические процессы, такие как обмен веществ, пролиферация, дифференцировка, апоптоз клеток. Отражена ассоциация некоторых заболеваний с изменением содержания микроРНК в периферическом русле крови. В статье представлена краткая характеристика группы некодирующих РНК. Дана характеристика основных баз данных микроРНК с отображением электронных адресов. Для определения микроРНК используют как традиционные методы, основанные на технологии амплификации, так и новые методы детекции (секвенирование нового поколения, электрохимическое детектирование на основе усиления ферментатного сигнала, идентификация с помощью лигирования и применения золотых наночастиц). Авторы проводят сравнения различных методов детекции микроРНК. Отмечено, что надэкспрессия или ингибирование генерации специфических микроРНК сопровождаются нарушениями важнейших функций печени и развитием заболеваний пищеварительной системы. Продемонстрировано, что изменения некоторых микроРНК в сыворотке крови или ткани печени являются высокодиагностическими маркерами некоторых заболеваний печени. Таким образом, идентификация изменения уровня представительности определенных микроРНК может дать ценную диагностическую информацию практикующему врачу, а влияние на процессы образования и матурации микроРНК при помощи лекарственных средств представляет собой новое направление терапии широкого спектра заболеваний. Особый интерес вызывает современное представление о диагностическом значении микроРНК при заболеваниях билиарного тракта у детей и возможностях медикаментозного управления активностью процесса их генерации.
The scientific review presents the significance of the world of microRNAs of the hepatobiliary system. For writing the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. It is emphasized that microRNAs in cells play an important role in regulating the activity of gene expression and control numerous physiological processes, such as metabolism, proliferation, differentiation, apoptosis of cells. The association of some diseases with changes in the content of microRNAs in the peripheral bloodstream is shown. The article presents a brief description of the group of non-coding RNAs. The characteristic of the basic microRNA databases with display of electronic addresses is given. Both traditional methods based on amplification technology and new detection methods (next-generation sequencing, electrochemical detection based on enzyme signal amplification, identification by ligation and application of gold nanoparticles) are used to determine microRNAs. The authors compare different methods of microRNA detection. It is noted that overexpression or inhibition of the generation of specific microRNAs is accompanied by impaired liver function and the development of diseases of the hepatobiliary system. Changes in some microRNAs in serum or liver tissue have been shown to be highly diagnostic markers of some liver diseases. Thus, the identification of changes in the level of representativeness of certain microRNAs may have valuable diagnostic information to the practitioner, and the impact on the processes of formation and maturation of microRNAs by drugs is a new direction in the treatment of a wide range of diseases. The modern idea of the diagnostic value of microRNAs in diseases of the biliary tract in children and the possibilities of drug management of the activity of the process of their generation are of particular interest.
геном; мікроРНК; методи детекції; гепатобіліарна система; огляд
геном; микроРНК; методы детекции; гепатобилиарная система; обзор
genome; microRNA; detection methods; hepatobiliary system; review
Вступ
Коротка характеристика групи некодуючих РНК
Бази даних мікроРНК
Методи детекції мікроРНК
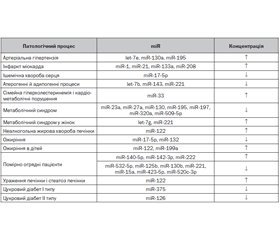
MiR-транскриптома гепатобіліарної системи
Висновок
- Абатуров О.Є., Бабич В.Л. Роль мікро-РНК при захворюваннях біліарної системи. Здоровье ребенка. 2017. № 7(12). С. 155-161. doi: 10.22141/2224-0551.12.7.2017.116191.
- Федянин М.Ю., Игнатова Е.О., Тюляндин С.А. Роль микро-РНК при солидных опухолях. Злокачественные опухоли. 2015. C. 3-14.
- Ambros V. The functions of animal microRNAs. Nature. 2004, Sep 16. 431(7006). 350-355. doi: 10.1038/nature02871.
- Backes C., Meder В., Hart М. et al. Prioritizing and selecting likely novel miRNAs from NGS data. Nucleic Acids Res. 2016. 44(6). e53. doi: 10.1093/nar/gkv1335.
- Bartel D.P. Metazoan MicroRNAs. Cell. 2018. 173. 20-51. https://doi.org/10.1016/j.cell.2018.03.006.
- Bertoli G., Cava C., Castiglioni I. MicroRNAs: New Biomarkers for Diagnosis, Prognosis, Therapy Prediction and Therapeutic Tools for Breast Cancer. Theranostics. 2015, Jul 13. 5(10). 1122-1143. doi: 10.7150/thno.11543.
- Budak H., Bulut R., Kantar M., Alptekin B. MicroRNA nomenclature and the need for a revised naming prescription. Brief. Funct. Genomics. 2016 Jan. 15(1). 65-71. doi: 10.1093/bfgp/elv026.
- Cammaerts S., Strazisar М., De Rijk P., Del J. Favero Genetic variants in microRNA genes: impact on microRNA expression, function, and disease. Front. Genet. 2015, May 21. 6. 186. doi: 10.3389/fgene.2015.00186.
- Cao B., Zhou X., Ma J. et al. Role of MiRNAs in Inflammatory Bowel Disease. Dig. Dis. Sci. 2017 Jun. 62(6). 1426-1438. doi: 10.1007/s10620-017-4567-1.
- Cao Y., Fanning S., Proos S. et al. A Review on the Applications of Next Generation Sequencing Technologies as Applied to Food-Related Microbiome Studies. Front. Microbiol. 2017, Sep 21. 8. 1829. doi: 10.3389/fmicb.2017.
- Cech T.R., Steitz J.A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 2014, Mar 27. 157(1). 77-94. doi: 10.1016/j.cell.2014.03.008.
- Chen J., Hu C., Pan P. Extracellular Vesicle MicroRNA Transfer in Lung Diseases. Front. Physiol. 2017, Dec 12. 8. 1028. doi: 10.3389/fphys.2017.01028.
- Crawford N.P. Deciphering the Dark Matter of Complex Genetic Inheritance. Cell. Syst. 2016, Mar 23. 2(3). 144-146. doi: 10.1016/j.cels.2016.03.003.
- Delihas N. Discovery and characterization of the first non-coding RNA that regulates gene expression, micF RNA: A historical perspective. World J. Biol. Chem. 2015, Nov 26. 6(4). 272-280. doi: 10.4331/wjbc.v6.i4.272.
- Dweep H., Gretz N. miRWalk2.0: a comprehensive atlas of microRNA-target interactions. Nat. Methods. 2015 Aug. 12(8). 697. doi: 10.1038/nmeth.3485.
- ENCODE Project Consortium An integrated encyclopedia of DNA elements in the human genome. Nature. 2012, Sep 6. 489(7414). 57-74. doi: 10.1038/nature11247.
- Filipowicz W., Bhattacharyya S.N., Sonenberg N. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight? Nat. Rev. Genet. 2008 Feb. 9(2). 102-114. doi: 10.1038/nrg2290.
- Finch M.L., Marquardt J.U., Yeoh G.C., Callus B.A. Regulation of microRNAs and their role in liver development, regeneration and disease. Int. J. Biochem. Cell. Biol. 2014 Sep. 54. 288-303. doi: 10.1016/j.biocel.2014.04.002.
- Iacomino G., Siani A. Role of microRNAs in obesity and obesity-related diseases. Genes. Nutr. 2017, Sep 25. 12. 23. doi: 10.1186/s12263-017-0577-z.
- Ichii O., Horino T. MicroRNAs associated with the development of kidney diseases in humans and animals. J. Toxicol. Pathol. 2018 Jan. 31(1). 23-34. doi: 10.1293/tox.2017-0051.
- Jeona T.-Il. Osborneb T.F. miRNA and cholesterol homeostasis. Biochim. Biophys. Acta. 2016 Dec. 1861(12 Pt B). 2041-2046. doi: 10.1016/j.bbalip.2016.01.005.
- Lee R., Feinbaum R., Ambros V. A short history of a short RNA. Cell. 2004, Jan 23. 116(2 Suppl). Р. 89-92, 1 p following S96. PMID: 15055592.
- Lee R.C., Feinbaum R.L., Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993, Dec 3. 75(5). 843-854. PMID: 8252621.
- Letelier P., Riquelme I., Hernández A.H. Circulating MicroRNAs as Biomarkers in Biliary Tract Cancers. Int. J. Mol. Sci. 2016, May 23. 17(5). pii: E791. doi: 10.3390/ijms17050791.
- Liu K., Tong H., Li T., Wang X., Chen Y. Research progress in molecular biology related quantitative methods of MicroRNA. Am. J. Transl. Res. 2020, Jul 15. 12(7). 3198-3211. PMID: 32774694; PMCID: PMC7407681.
- Londin E., Loher P., Telonis A.G. et al. Analysis of 13 cell types reveals evidence for the expression of numerous novel primate- and tissue-specific microRNAs. Proc. Natl Acad. Sci. USA. 2015, Mar 10. 112(10). 1106-1115. doi: 10.1073/pnas.1420955112.
- Marian A.J., van Rooij E., Roberts R. Genetics and genomics of single-gene cardiovascular diseases: common hereditary cardiomyopathies as prototypes of single-gene disorders. Journal of the American College of Cardiology. 2016. 68(25). 2831-2849. doi: 10.1016/j.jacc.2016.09.968.
- McCall M.N., Kim M.S., Adil M. et al.Toward the human cellular microRNAome. Genome Res. 2017. 27(10). 1769-1781. doi: 10.1101/gr.222067.117.
- McGeary S.E., Lin K.S., Shi Ch.Y. The biochemical basis of microRNA targeting efficacy. Science. 2019. 366(6472). aav1741. doi: 10.1126/science.aav1741.
- Mellis D., Caporali A. MicroRNA-based therapeutics in cardiovascular disease: screening and delivery to the target. Biochem. Soc. Trans. 2018, Feb 19. 46(1). 11-21. doi: 10.1042/BST20170037.
- Meyts I., Bosch B., Bolze A. et al. Exome and genome sequencing for inborn errors of immunity. J. Allergy Clin. Immunol. 2016 Oct. 138(4). 957-969. doi: 10.1016/j.jaci.2016.08.003.
- Minatel B.C., Martinez V.D., Ng K.W. et al. Large-scale discovery of previously undetected microRNAs specific to human liver. Hum. Genomics. 2018, Mar 27. 12(1). 16. doi: 10.1186/s40246-018-0148-4.
- Mingardi J., Musazzi L., De Petro G., Barbon A. miRNA Editing: New Insights into the Fast Control of Gene Expression in Health and Disease. Mol. Neurobiol. 2018, Feb 19. doi: 10.1007/s12035-018-0951-x.
- Mitchell P.S., Parkin R.K., Kroh E.M. et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proc. Natl Acad. Sci. USA. 2008, Jul 29. 105(30). 10513-10518. doi: 10.1073/pnas.0804549105.
- Panzeri I., Rossetti G., Abrignani S., Pagani M. Long Intergenic Non-Coding RNAs: Novel Drivers of Human Lymphocyte Differentiation. Front. Immunol. 2015, Apr 15. 6. 175. doi: 10.3389/fimmu.2015.00175.
- Papanagnou P., Stivarou T., Tsironi M. The Role of miRNAs in Common Inflammatory Arthropathies: Osteoarthritis and Gouty Arthritis. Biomolecules. 2016, Nov 11. 6(4). pii: E44. doi: 10.3390/biom6040044.
- Picard C., Fischer A. Contribution of high-throughput DNA sequencing to the study of primary immunodeficiencies. Eur. J. Immunol. 2014 Oct. 44(10). 2854-2861. doi: 10.1002/eji.201444669.
- Piriyapongsa J., Bootchai С., Ngamphiw С., Tongsima S. microPIR2: a comprehensive database for human-mouse comparative study of microRNA-promoter interactions. J. Piriyapongsa. Database (Oxford). 2014, Nov 25. 2014. bau115. doi: 10.1093/database/bau115.
- Ramassone A., Pagotto S., Veronese A., Visone R. Epigenetics and MicroRNAs in Cancer. Int. J. Mol. Sci. 2018, Feb 3. 19(2). pii: E459. doi: 10.3390/ijms19020459.
- Seleman M., Hoyos-Bachiloglu R., Geha R.S., Chou J. Uses of Next-Generation Sequencing Technologies for the Diagnosis of Primary Immunodeficiencies. Front. Immunol. 2017, Jul 24. 8. 847. doi: 10.3389/fimmu.2017.00847.
- Siggens L., Ekwall K. Epigenetics, chromatin and genome organization: recent advances from the ENCODE project. J. Intern. Med. 2014 Sep. 276(3). 201-214. doi: 10.1111/joim.12231.
- Signal B., Gloss B.S., Dinger M.E. Computational Approaches for Functional Prediction and Characterisation of Long Noncoding RNAs. Trends Genet. 2016 Oct. 32(10). 620-637. doi: 10.1016/j.tig.2016.08.004.
- Takata A., Otsuka M., Yoshikawa T. et al. MicroRNAs and liver function. Minerva Gastroenterol. Dietol. 2013 Jun. 59(2). 187-203. PMID: 23831909.
- Turchinovich A., Weiz L., Langheinz A., Burwinkel B. Characterization of extracellular circulating microRNA. Nucleic. Acids Res. 2011, Sep 1. 39(16). 7223-7233. doi: 10.1093/nar/gkr254.
- Valinezhad Orang A., Safaralizadeh R., Kazemzadeh-Bavili M. Mechanisms of miRNA-Mediated Gene Regulation from Common Downregulation to mRNA-Specific Upregulation. Int. J. Genomics. 2014. 2014. 970607. doi: 10.1155/2014/970607.
- Wang X., He Y., Mackowiak B., Gao B. MicroRNAs as regulators, biomarkers and therapeutic targets in liver diseases. Gut. 2020, Oct 30: gutjnl-2020-322526. doi: 10.1136/gutjnl-2020-322526. Epub. ahead of print. PMID: 33127832.
- Wang Y., Xu D., Wang B., Hou X. Could MicroRNAs be Regulators of Gout Pathogenesis? Cell. Physiol. Biochem. 2015. 36(6). 2085-2092. doi: 10.1159/000430176.
- Wasik U., Kempinska-Podhorodecka A., Bogdanos D.P. et al. Enhanced expression of miR-21 and miR-150 is a feature of anti-mitochondrial antibody-negative primary biliary cholangitis. Mol. Med. 2020. 26(8). https://doi.org/10.1186/s10020-019-0130-1.
- Weber J.A., Baxter D.H., Zhang S. et al. The microRNA spectrum in 12 body fluids. Clin. Chem. 2010 Nov. 56(11). 1733-1741. doi: 10.1373/clinchem.2010.147405.
- Willeit P., Skroblin P., Kiechl S. et al. Liver microRNAs: potential mediators and biomarkers for metabolic and cardiovascular disease? Eur. Heart J. 2016, Nov 14. 37(43). 3260-3266. doi: 10.1093/eurheartj/ehw146.
- Yin X., Chai Z., Sun X., Chen J. et al. Overexpression of microRNA-96 is associated with poor prognosis and promotes proliferation, migration and invasion in cholangiocarcinoma cells via MTSS1. Experimental and Therapeutic Medicine. 2020. 4(19). 2757-2765. https://doi.org/10.3892/etm.2020.8502.
- Zhang T., Yang Z., Kusumanchi P., Han S., Liangpunsakul S. Critical Role of microRNA-21 in the Pathogenesis of Liver Diseases. Front. Med. 2020. 7. 7. doi: 10.3389/fmed.2020.00007.

/123.jpg)
/124.jpg)
/125.jpg)
/126.jpg)
/127.jpg)
/128.jpg)
/129.jpg)